EmbryoNet: Entwicklungsschäden automatisiert erkennen
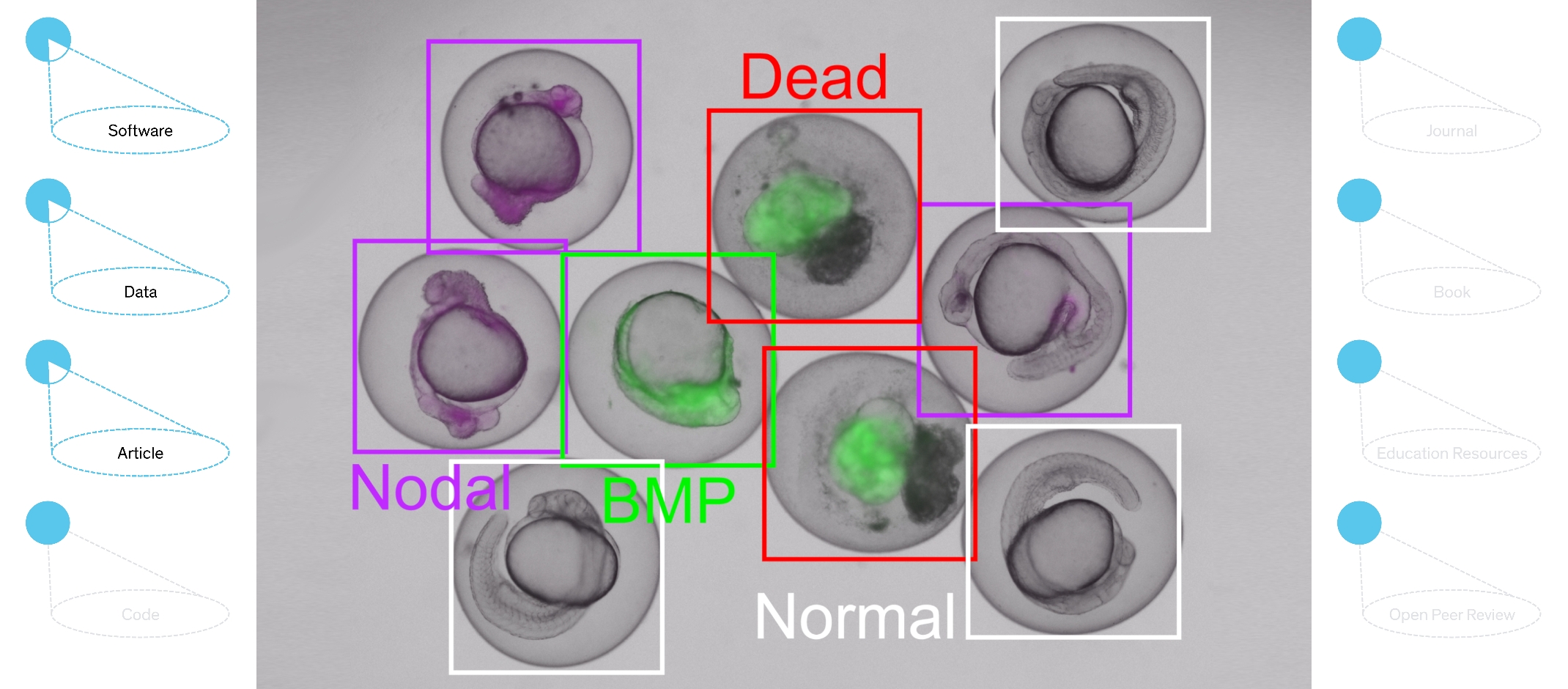
Damit aus einer befruchteten Eizelle ein komplexer, mehrzelliger Organismus entstehen kann, ist der Ablauf der Embryonalentwicklung biologisch genauestens reguliert. Hierbei spielen eine Reihe von Signalwegen eine entscheidende Rolle. Werden die Signalwege in ihrer Aktivität gestört oder unterbrochen, führt dies zu charakteristischen Entwicklungsschäden beim Embryo.
Auf Bildmaterial erkennt und klassifiziert die Open-Source-Software EmbryoNet diese Schäden bei Tieren dank künstlicher Intelligenz automatisiert. Anhand der Klassifizierung können anschließend Rückschlüsse auf Störungen der für die embryonale Entwicklung wichtigsten Signalwege gezogen werden. Dass die Software effektiver und genauer arbeitet als menschliche ExpertInnen demonstrieren die Entwickler um Patrick Müller, Professor für Entwicklungsbiologie an der Universität Konstanz, am Beispiel von Zebrafisch-Embryonen.
Die Open-Source-Software EmbryoNet kann kostenlos aus dem GitHub-Verzeichnis der Arbeitsgruppe Patrick Müller heruntergeladen werden. Zusätzlich bieten die Entwickler in ihrer Cloud eine Installationsdatei für Windows-Rechner zum Download an.
Die Trainings- und Testdaten für EmbryoNet (doi: 10.48606/15) finden Sie auf KonDATA zum Download.
Den Begleitartikel zur Software in Nature Methods (doi: 10.1038/s41592-023-01873-4) finden Sie kostenlos zugänglich auf dem Konstanzer Online-Publikationssystem (KOPS).