Das Mikroskop für Proteinstrukturen

Eine neue High-Tech-Methode der Mikroskopie ist aussichtsreich, um Schlüsselvorgänge des Lebens auf molekularer Ebene aufzuklären: Die sogenannte Spin-basierte Quanten-Lichtmikroskopie (SQLM) kombiniert bestehende Analyseverfahren und könnte es dadurch ermöglichen, ein komplexeres Bild von den biologischen Mechanismen von Proteinen in der lebenden Zelle zu gewinnen. Ein Forschungsverbund an der Universität Konstanz wird nun von der Deutschen Forschungsgemeinschaft (DFG) mit 1,3 Millionen Euro dabei unterstützt, die SQLM im Bereich der Strukturbiologie und Biophysik in die Anwendung zu bringen. Die DFG stellt den Förderbetrag zur Anschaffung des High-Tech-Mikroskops in Konstanz zur Verfügung. Die Technologie bildet die Grundlage für angehende Spitzenforschungsprojekte im Bereich der Lebenswissenschaften.
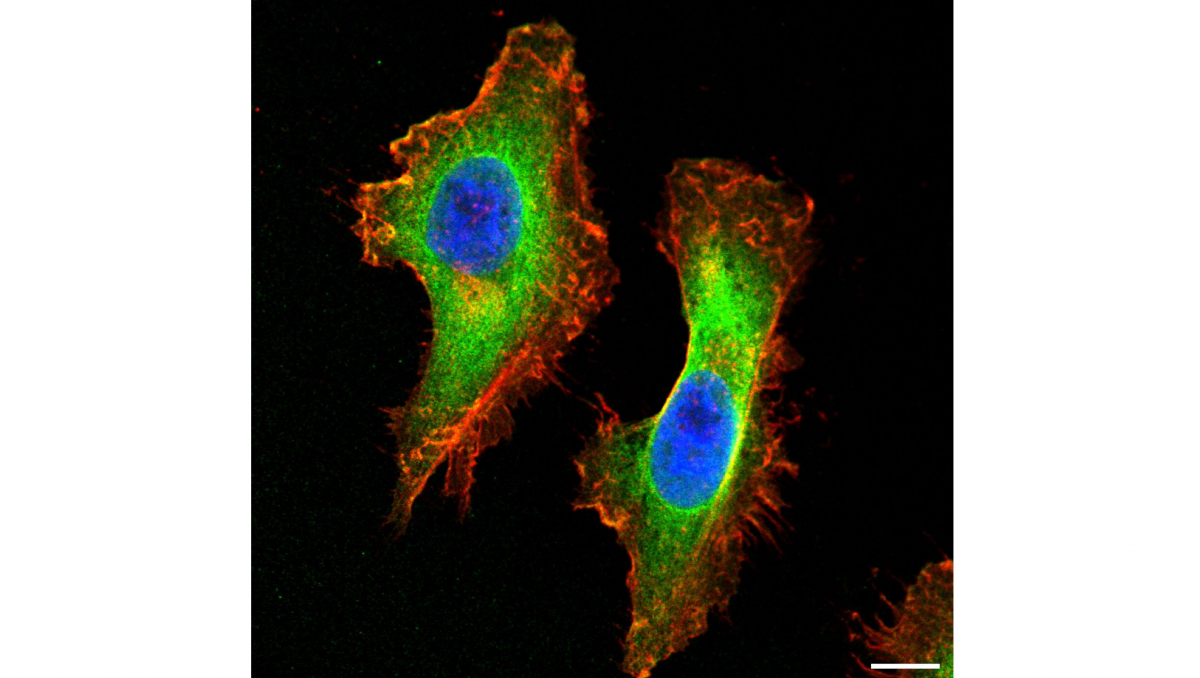
Wer die Geheimnisse des Lebens entschlüsseln will, muss sehr genau hinschauen. Zum Beispiel bei Proteinen: Sie sind Grundbausteine des Lebens, doch ihre Struktur und ihr Zusammenspiel mit den weiteren Bausteinen des Körpers sind mit dem bloßen Auge nicht erkennbar. In der Praxis findet ihre Untersuchung typischerweise im Reagenzglas (in vitro) statt, mit Hilfe von fortgeschrittenen Analysetechniken. An der Universität Konstanz kommen hierfür unter anderem die Magnetische Resonanzspektroskopie sowie hochentwickelte Lichtmikroskopie zum Einsatz. Beide Verfahren haben ihre Stärken und ihre spezifischen Anwendungsgebiete – doch beide zeigen jeweils nur einen Teil des Gesamtbilds.
„In der Spin-basierten Quanten-Lichtmikroskopie wollen wir nun beide Verfahren miteinander kombinieren“, schildert Prof. Dr. Malte Drescher, Sprecher des Forschungsverbunds und Prorektor für Forschung an der Universität Konstanz. Das neue Verfahren vereint zwei bislang getrennte Perspektiven auf denselben Gegenstand und liefert damit ein umfassenderes Gesamtbild der untersuchten Prozesse.
Die Anwendung von SQLM auf mikrobiologische Strukturen ist neu:
© Inka Reiter„Unser langfristiges Ziel ist, mittels der SQLM sowohl die Struktur von Proteinen als auch deren Positionierung, Dynamik und Interaktionen gleichzeitig beobachten zu können – und das in der lebenden Zelle und bei Raumtemperatur, was bislang in dieser Form nicht möglich war.“
Prof. Dr. Malte Drescher, Sprecher des Forschungsverbunds, Prorektor für Forschung an der Universität Konstanz
Der Chemiker und Experte für die Spektroskopie komplexer Systeme ist sich sicher: „Als ‚Mikroskop für Proteinstrukturen‘ hat die Spin-basierte Quanten-Lichtmikroskopie das Potenzial, unser Verständnis der Proteostase und der zellulären Adaption weit über den heutigen Forschungsstand hinaus voranzutreiben.“
Zwei Perspektiven vereint: Wie SQLM funktioniert
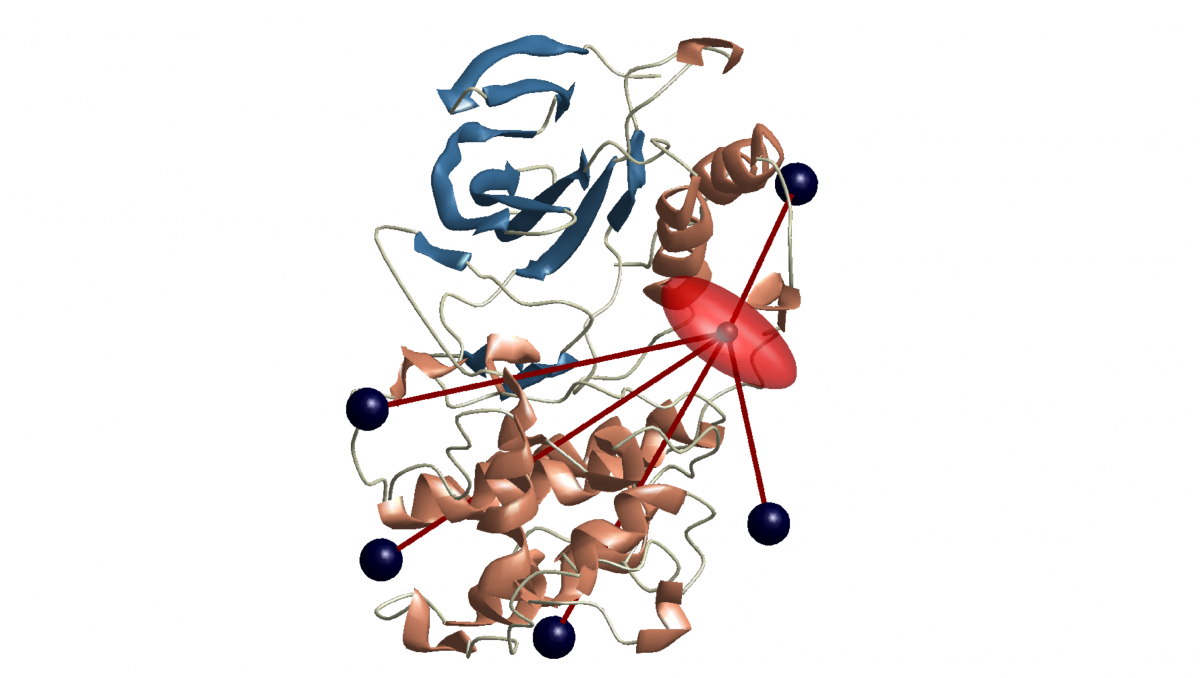
Die Spin-basierte Quanten-Lichtmikroskopie verbindet die Lichtmikroskopie, die detaillierte Informationen über die Lage von Molekülen liefert, mit der Magnetischen Resonanzspektroskopie, die Aufschluss über die Struktur der Moleküle gibt (z.B. die Faltung von Proteinen). Damit vereint SQLM zwei Perspektiven, die bislang separat eingeholt werden mussten, und liefert damit ein Gesamtbild von Struktur, Position und Dynamik der untersuchten Moleküle.
Vereinfacht gesagt nutzt SQLM „optisch detektierte Magnetresonanz“, um Informationen über Moleküle auf Nanometer-Ebene (der tausendste Teil eines Mikrometers) zu gewinnen. Wir können uns das kombinierte Verfahren wie folgt vorstellen: Von der untersuchten Zelle wird eine Mikroskop-Aufnahme erstellt. Anders als bei der klassischen Lichtmikroskopie können wir hier nun aber einen Bildpunkt herausgreifen und an diesem gleichzeitig eine Magnetische Resonanzspektroskopie durchführen. Die Spektroskopie verrät uns die Struktur der Moleküle an diesem Bildpunkt, während die Lichtmikroskopie uns zugleich einen Überblick über deren Positionierung gibt. Eine besondere Rolle spielen dabei sogenannte Spin-Marker: Die Quantenzustände von Elektronenspins werden bei der Spektroskopie als Detektoren eingesetzt und ermöglichen die präzise Abstandsmessung auf Nanometer-Ebene.
Der Schritt in die wissenschaftliche Anwendung
SQLM wurde in den vergangenen Jahren zur technischen Reife gebracht, jetzt soll das Verfahren in der Wissenschaft zur Anwendung kommen. Der Konstanzer Forschungsverbund sieht ein besonders hohes Potenzial von SQLM bei der Analyse von mikrobiologischen Strukturen, insbesondere Proteinen. Zu den Schlüsselfragen, die das Team aus Konstanz mithilfe der SQLM beantworten möchte, zählen:
-
Wie hängt die Faltung von intrinsisch ungeordneten Proteinen mit ihrer Position in der Zelle zusammen?
-
Wie fördern molekulare Chaperone die korrekte Faltung von anderen Proteinen in der lebenden Zelle?
-
Wie führen fehlerhaft gefaltete oder beschädigte Proteine zu neurodegenerativen Krankheiten?
Hierzu möchte der Konstanzer Forschungsverbund die Spin-basierte Quanten-Lichtmikroskopie methodisch weiterentwickeln und im Bereich der Strukturbiologie und Biophysik in die Anwendung bringen. In dem Forschungsverbund kommen Kompetenzen unter anderem aus der Molekularen Mikrobiologie, der Biochemie, Spektroskopie, Quantenoptik, Photonik sowie Chemoinformatik zusammen. Neben der Universität Konstanz ist auch eine Arbeitsgruppe der Universität Ulm beteiligt.
Gebündelte Kompetenzen, geteilte Infrastruktur
Ein Meilenstein des Projekts wird der Aufbau einer neuen Core Facility SQLM an der Universität Konstanz sein, also eines Infrastruktur- und zugleich Kompetenzzentrums im Bereich der neuen SQLM-Methode. In diesem Zentrum wird einerseits die Technologie gebündelt und kommen andererseits die Expertisen aus den beteiligten Fachbereichen zusammen.
Gemäß des Konstanzer Modells der Core Facilities ist dies eine geteilte Infrastruktur. Sprich: Jede Wissenschaftlerin und jeder Wissenschaftler der Universität Konstanz hat Zugriff darauf – und auch Externe können die Einrichtung auf Anfrage nutzen. Die geplante Core Facility unterstützt den Forschungsschwerpunkt „Chemische Biologie“ an der Universität Konstanz.